影像组学如何实际操作?
近年来,医学领域迅猛发展,促进了医学影像技术的持续进步,随之产生了大量的影像数据。在此背景下,影像组学作为一种针对大规模医学影像数据的研究方法应运而生。随着影像组学在临床诊断和治疗中的广泛应用,如何迅速且高效地复现影像组学流程,已成为众多研究者关注的热点问题。英瑞云自主研发英瑞云MedAI平台,无需编程,用户可借助内置工具轻松复现影像组学研究论文中的相关流程。
平台一站式解决了医学影像大数据在存储、共享、脱敏、格式转换、显示、数据标准化、人工智能计算、智能标注、成果与数据共享以及交叉学科团队协作等方面的难题,极大地消除了医学AI开放创新的阻碍。此外,平台还提供了便捷的影像处理工具,实现了零代码的特征工程训练,是医生们的智能化的实验辅助伙伴。

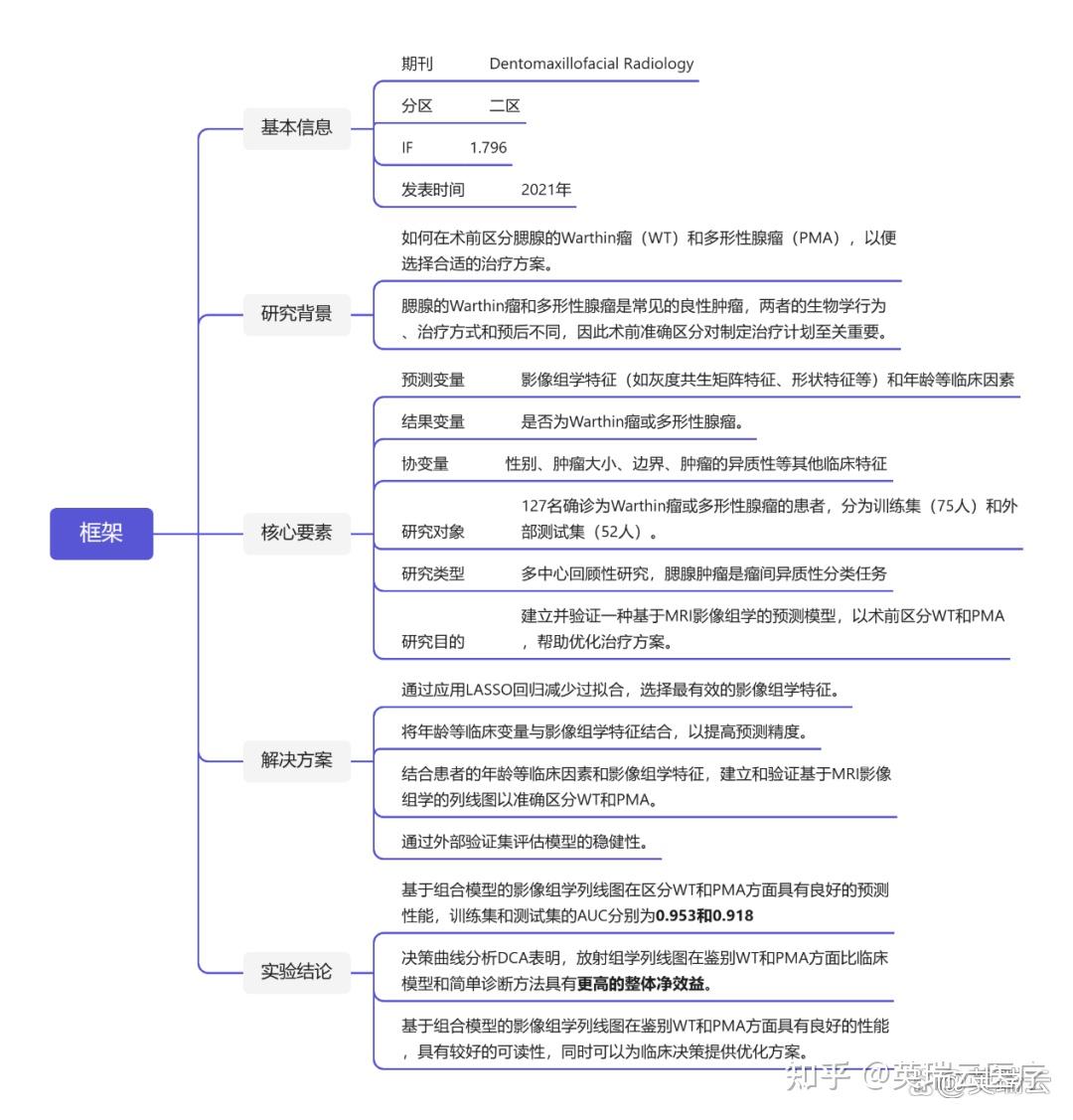
本期选择的影像组学复现论文是发表在DentoMaxilloFacial Radiology杂志的《Development and validation of an MRI-based radiomics nomogram for distinguishing Warthin's tumour from pleomorphic adenomas of the parotid gland》,该论文是一项针对术前区分腮腺Warthin瘤(WT)与多形性腺瘤(PMA)以支持治疗决策的研究。
该文章主要内容是对腮腺肿瘤(WT和PMA)的磁共振图像使用计算机手段从MR图像中提取高通量影像特征,临床与影像组学特征提取和筛选后,建立模型以术前区分WT还是PMA帮助治疗决策。
方法:
收集127例经病理证实的WT或PMA患者,分为训练集(75例)和外部测试集(52例),提取临床特征与影像组学特征并筛选后,构建三种不同特征的模型:独立临床特征logistic回归模型;影像组学特征logistic回归模型;影像组学组合独立临床特征的logistic回归模型。经过比较分析ROC曲线与决策曲线分析(DCA)发现组合模型的区分效能最好。最后根据组合模型构建影像组学列线图以增加预测模型的结果可读性。
提炼亮点:
1.首次将影像组学应用于鉴别WT和PMA;
2.构建三种模型比较分析,凸显影像组学特征的鉴别能力;
3.开发了一种基于MRI的影像组学列线图,用于术前鉴别WT和PMA,具有良好的预测性能。
精炼文章流程:
1.对临床特征进行logistic回归建模;
2.提取影像组学特征,通过lasso回归进行特征筛选再logistic回归建模;
3.结合影像组学特征+临床特征进行建模;
4.绘制三个模型的ROC曲线进行对比;
5.对组合模型绘制诺莫图+决策曲线+校准曲线。
这项研究的主体方法与亮点方法——logistic回归构建三模型比较分析,完全可以通过英瑞云影像组学平台进行复现。平台人工智能模块的影像组学功能提供对于不同模式下不同的影像组学研究方法,如下图,针对影像组学二分类任务的单模态单标签,即利用机器学习算法构建影像组学模型,以对样本进行类别预测。下边将针对文章主体方法进行复现帮助大家理解影像组学分析全流程以及学习平台使用方法以辅助研究。

复现流程
1、 关于数据
BraTS2019(Brain Tumor Segmentation Challenge 2019)数据集是专门用于脑肿瘤分割任务的公开医疗数据集,广泛用于医学图像处理和机器学习研究,尤其是在肿瘤的自动分割、分类、和定量分析等领域。
数据集内容
BraTS2019数据集包含335例经过预处理的脑肿瘤患者的磁共振成像 (MRI)数据,涵盖259例Hgg高级别胶质瘤和76例低级别胶质瘤。每个病例包括3D的MRI图像,提供了四种不同的模态:
1. T1 权重(T1-weighted)
2. T1Gd 对比增强(T1-weighted with gadolinium-enhancement)
3. T2 权重(T2-weighted)
4. FLAIR(Fluid Attenuated Inversion Recovery)
标签信息
数据集中有经过专家注释的肿瘤分割标签。肿瘤区域根据不同的病理特征被划分为以下几个部分:
1. 增强肿瘤区(Enhancing Tumor, ET),标注为1;
2. 肿瘤周围水肿区(Peritumoral Edema, ED),标注为2;
3. 非增强肿瘤核(Necrotic and Non-enhancing Tumor Core, NCR/NET),标注为4。
临床数据
数据集原始表格中提供了HGG患者的年龄数据,LGG患者没有提供年龄数据。
数据预处理
为了使读者更方便的体验我们的平台开展可视化实验,我们将四种模态的数据重新命名为image_XXX格式,具体映射如下:
t1---image_1
t1ce---image_2
flair---image_3
t2---image_4
此外,我们还对标签进行了预处理,将标签转换为二分类标签,具体说明如下:
mask_1: 增强肿瘤区
mask_2: 肿瘤周围水肿区
mask_3: 非增强肿瘤核
mask_4: 增强肿瘤区 + 肿瘤周围水肿区 + 非增强肿瘤核
最后,我们对临床表格进行了手动处理,删除了部分HGG患者,并为LGG患者生成 一个25-55之间的随机数作为年龄,最终保留了241例HGG患者和77例LGG患者。在任务列中,将HGG患者设置为1,LGG患者设置为2。预处理后的文件夹结构如下:

开始实验
step 1: 通过分享链接,将数据集文件保存至云空间。
step 2: 在平台上进入人工智能模块。

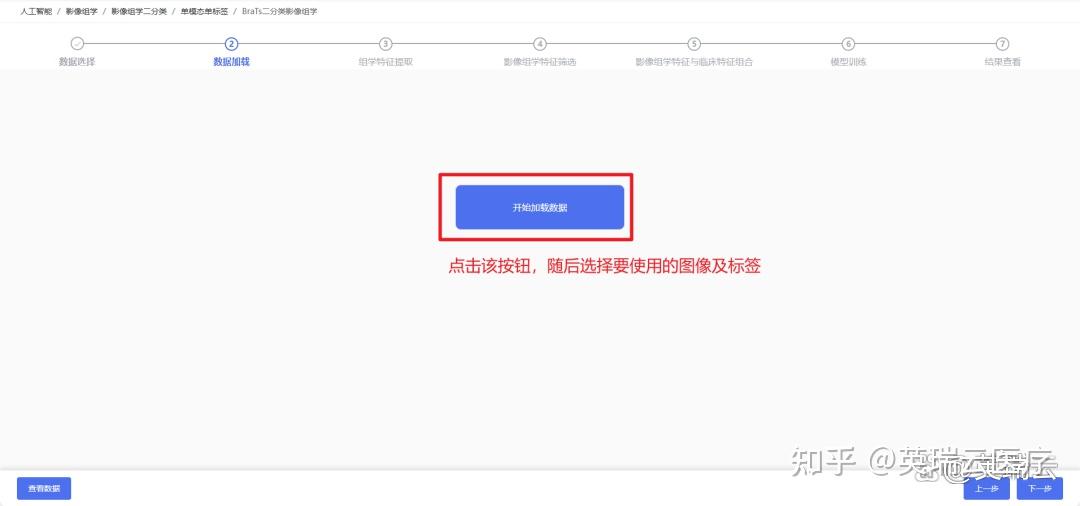
step 3:选择“影像组学二分类 - 单模态单标签”,输入任务名称并创建任务后,进入数据选择步骤。
step 4:在任务界面,选择数据并进行数据格式检查。

step 5:数据格式检查通过后,进行数据加载操作。在本例中,我们使用image_4.nii.gz 和 mask_4.nii.gz ,并将任务正类选择为1。
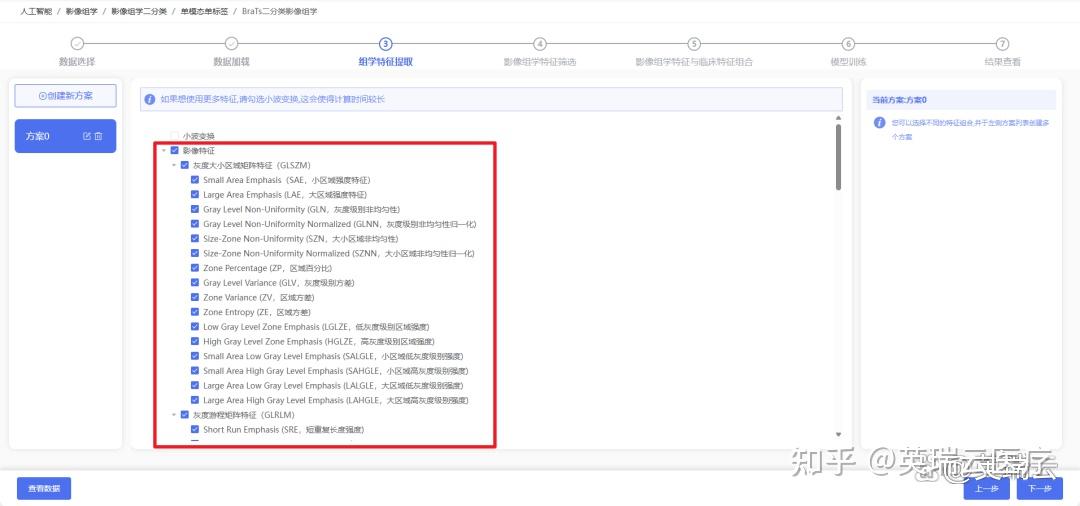
step 6:进行影像特征选择及要使用的特征提取方法,在本例中,我们选择全部的影像特征,并使用基于LASSO的特征筛选方法,等待计算完毕后,即可查看筛选出来的影像特征。


step 7:进行影像组学特征与临床特征组合操作,在该步骤中,可以选择多种组合方案,例如仅使用影像特征、仅使用临床特征、自选影像特征与自选临床特征组合等方案;在本例中,我们使用全部的影像特征和全部的临床特征。

step 8:最后,选择要使用的分类模型及参数,即可开始模型训练;在本例中, 我们使用逻辑回归方法。

待模型运行完毕后,即可查看多种类型的可视化结果,并支持一键生成结果报告。
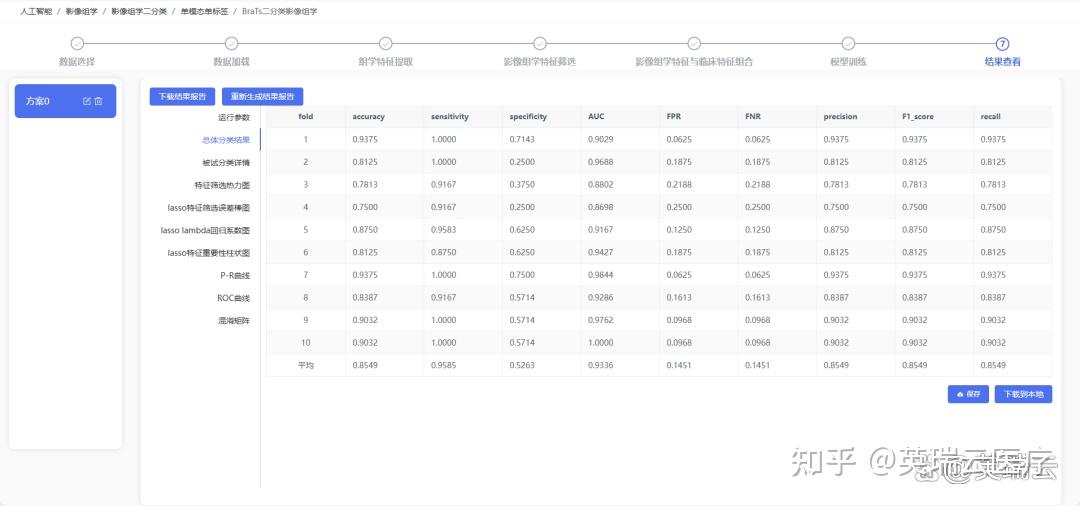
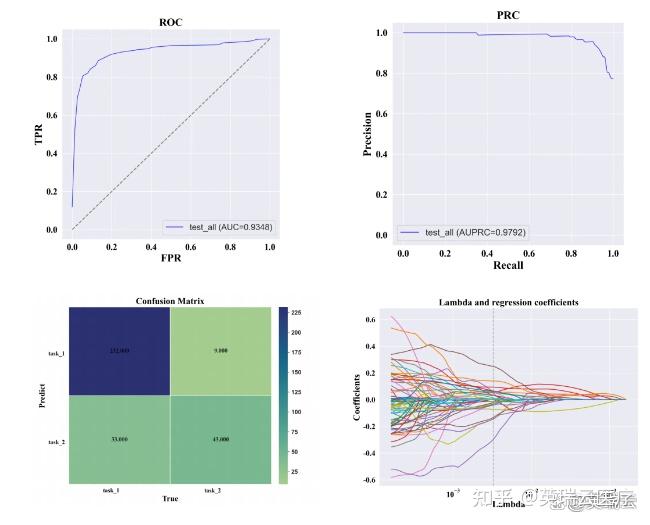
实验报告查看
 新公网安备 65010402001845号
新公网安备 65010402001845号