生物信息学入门需要具备什么能力?
首先,生信入门很简单,别被网上那些人说的吓到!
其次,个人认为,生信不比其他生物行业有多大优势。
下面简单说一说,生物信息学如何入门
嫌Matplotlib繁琐?试试Seaborn!10W字《Python可视化教程1.0》来了!10W字《R ggplot2可视化教程1.0》来了!了解一般的生物信息在干嘛
开局一张图,生物信息,生物开头,就是告诉您生物信息的终极目的还是在搞生物,是要解决生物问题,所以先要知道生物信息要用到哪些技术解决哪些问题,一个生信流程又是咋样的。
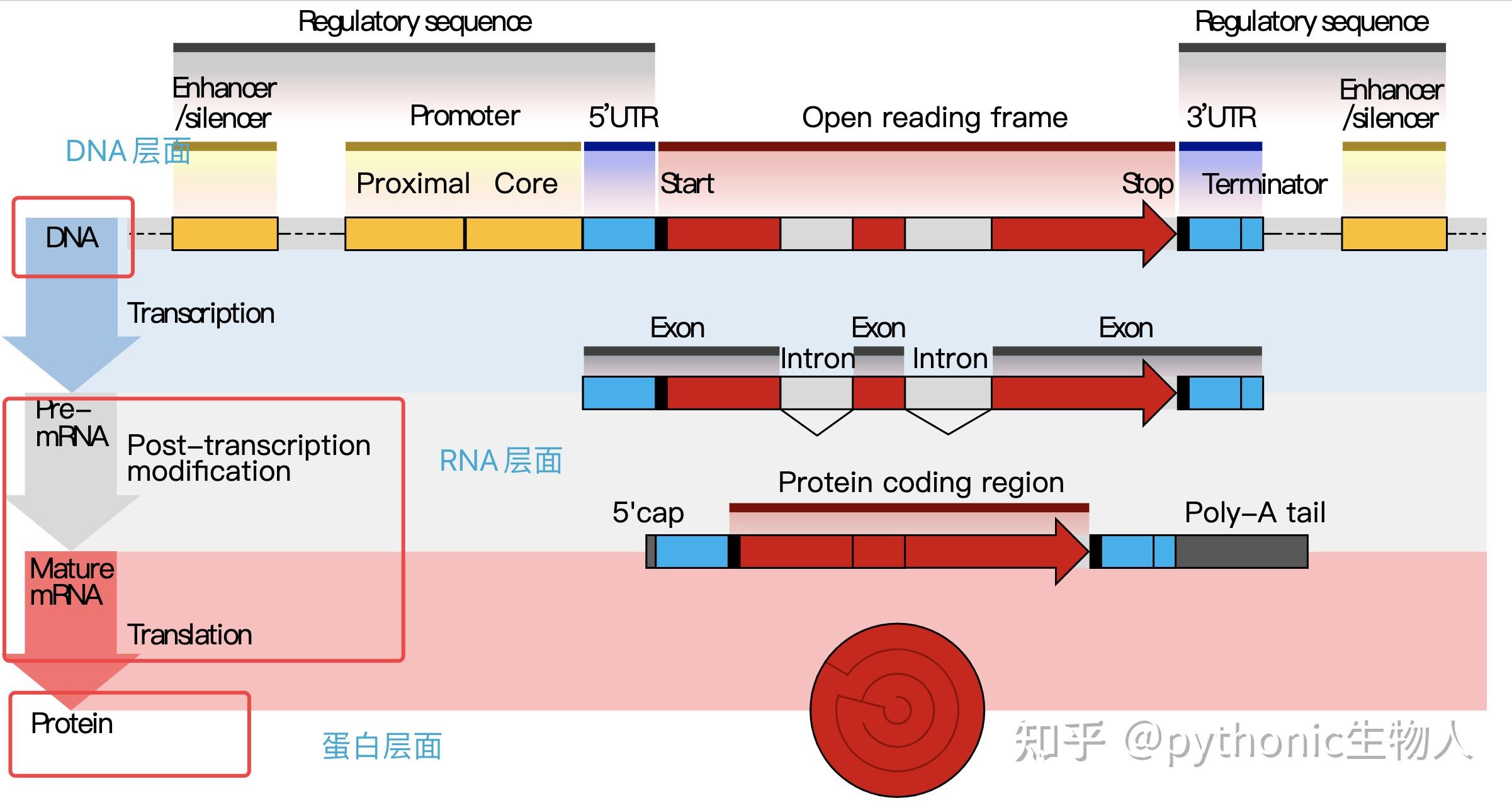
围绕上图DNA-RNA-蛋白的转录、翻译的生物过程,可浅显的理解生物信息是由DNA组、RNA组、蛋白组、代谢组等等各种组学组成。
了解DNA/RNA等组学
基本上擅长一种就可以去找到工作了。
RNA组学:可细分为mRNA|miRNA|lncRNA|circRNA等组学技术
还是一张图,可以一览各个RNAseq之间联系,

常规RNAseq分析都分析个啥?

前几年小编水过几篇文章,可以大致看看都在干什么:
- Omics精进09|mRNA生信分析常规流程
- Omics精进08|mRNA测序文库构建
- Omics精进07|转录组之mRNA|miRNA|lncRNA|circRNA测序简介
DNA层面:也可简单细分为WGS(Low-pass NIPT/NIPTPLUS/CNVseq、标准WGS)/WES/panel等技术。
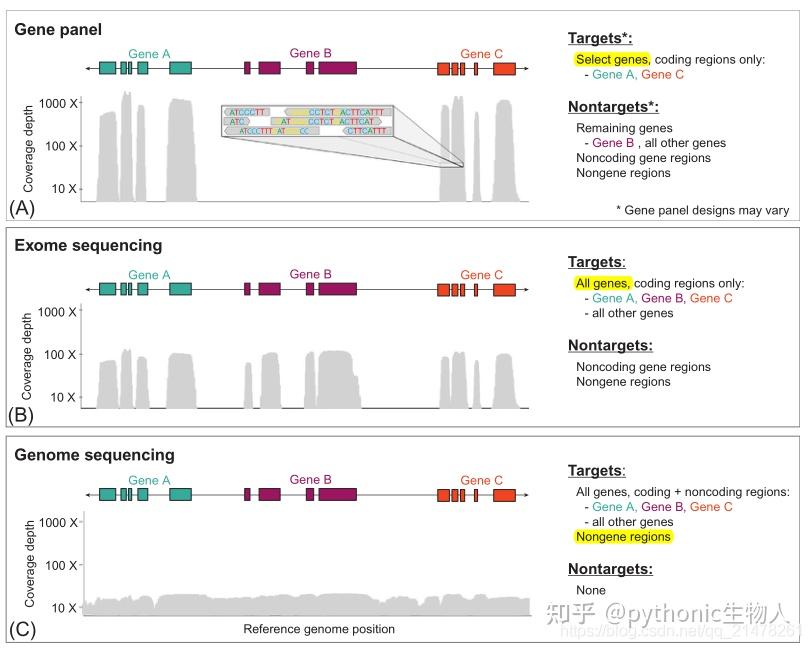
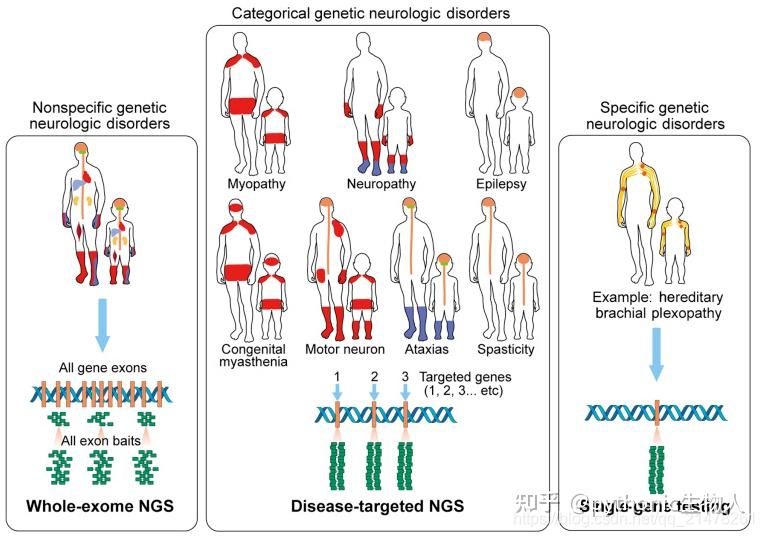
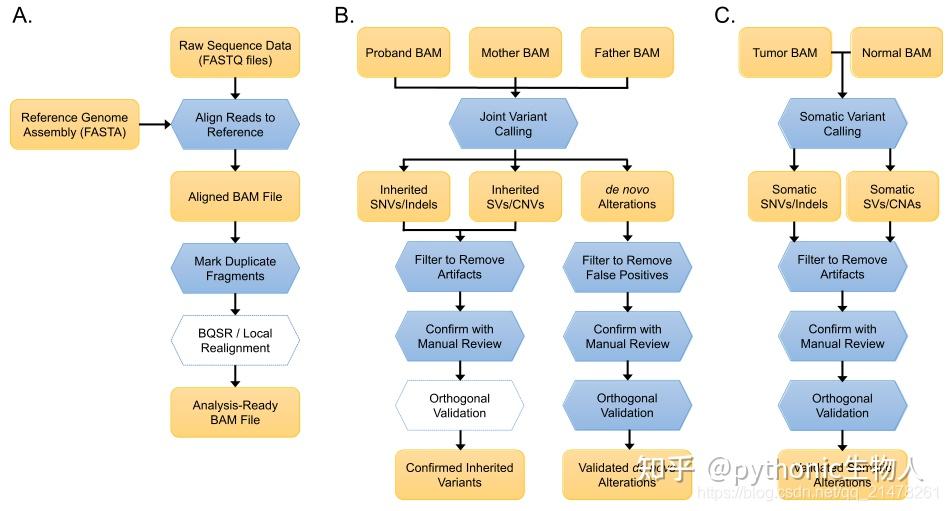
也可简单看看都在分析个啥?
- Omics精进06|Gene panel检测报告解读
- Omics精进02|临床Gene Panel设计
- Omics精进01|临床WGS/WES/Gene Panel/Single gene异同
上面简单介绍了各组学技术后,为了更好的分析,需要了解一下各个组学数据是怎么测序来的,知其然,才不会沦为“pipeliner”
了解测序原理
市面上大部分组学数据还是由欧美国家边合成边测序的仪器测序而来,可简单参考几篇水文:
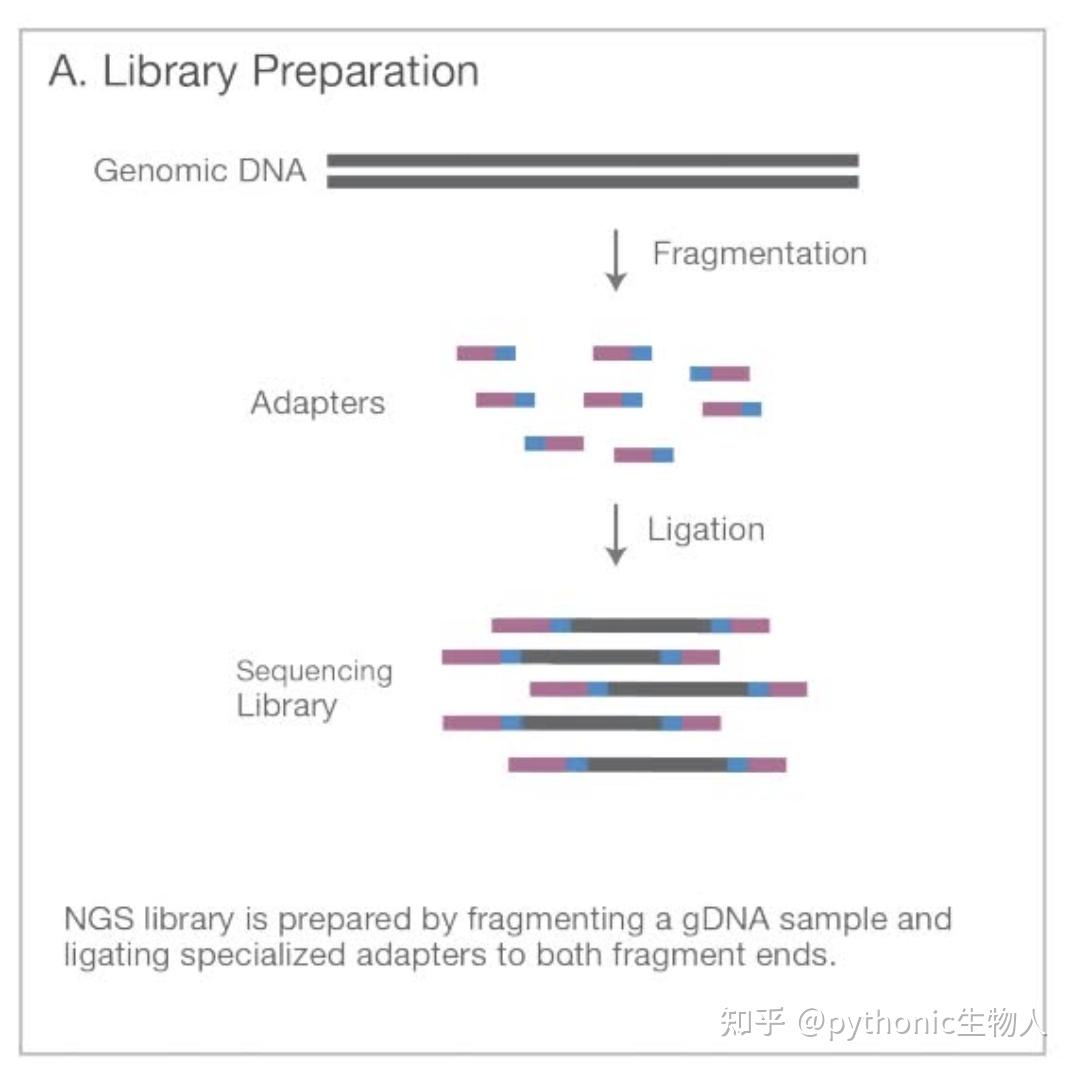
- NGS测序基础梳理01-文库构建
为目的DNA两边添加接头,以便与测序芯片flowcell结合,
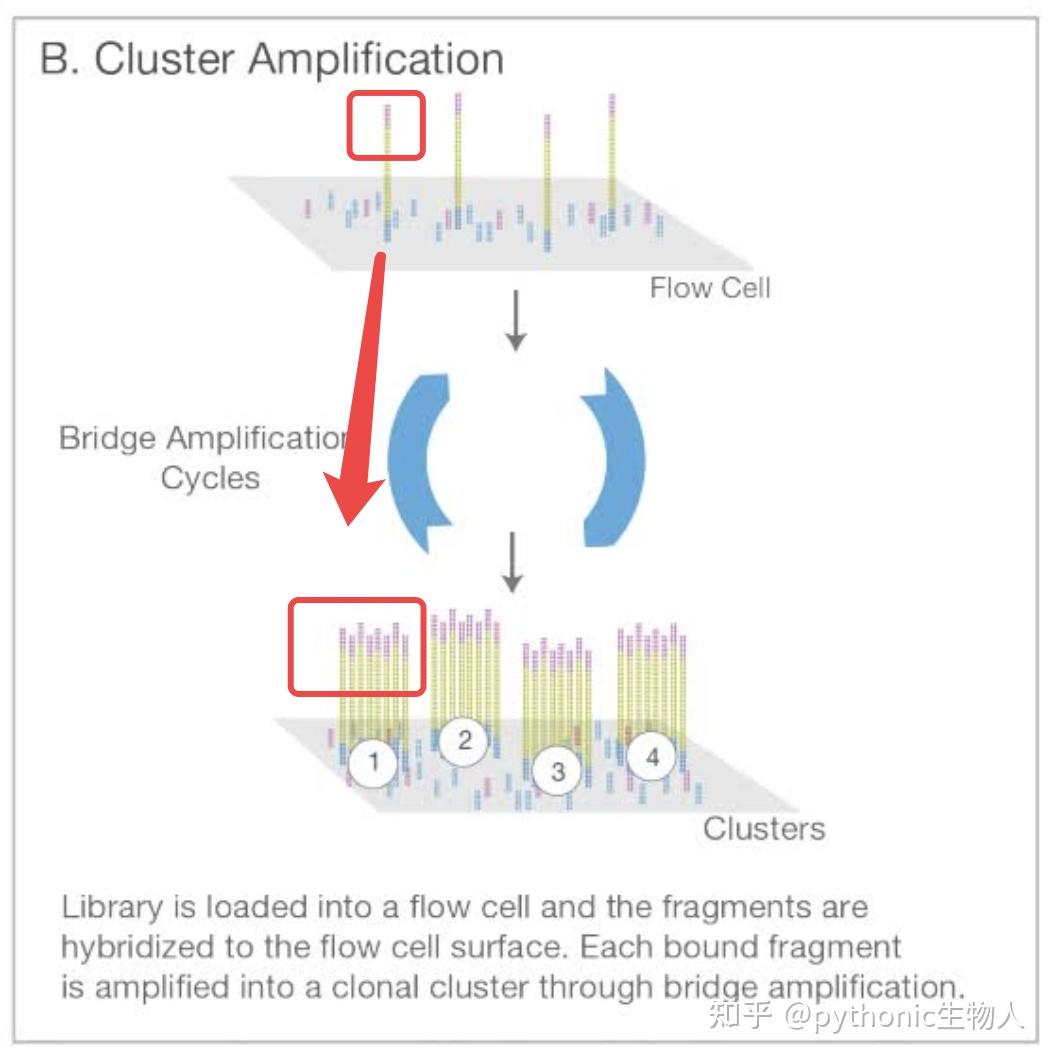
- NGS测序原理梳理02-簇生成
将上一步的文库与flowcell结合,扩增放大信号的过程,
- NGS测序原理梳理03-边合成边测序
以特殊处理的dNTP(处理后一次只能连接/合成上一个ATGC、发一种特定的光/测序)为材料,通过sbs测序,是一个化学信号--光信号-测序碱基的过程,

生信常见的数据格式
顺便看看生信常见的数据格式:

- NGS数据格式梳理02-SAM/BAM格式最详细解读
- NGS数据格式梳理01-FASTQ和FASTA格式详解
了解linux
生信分析计算量很大,测序数据一般都是N个G,必须上服务器,而这些服务器一般都是基予linux系统,要在上面操作,需要熟练常用的linux命令(就那么几个)
一定要看书的话,不用买:鸟哥的Linux私房菜简体首页
ln -s
less -NS
cat
tar
gunzip
awk/sed/grep三兄弟
cp
mv
rm -rf不要随便用
scp
wget/curl
cut
sort
uniq
find
top
xargs
等等。。。。
有小伙伴说我自己没服务器咋办,如果是mac的话,可以终端直接练习;
如果是windows可以捣鼓一下vmware,或者power shell。
了解生信软件安装
用conda啊、docker啊、或者源码安装等等,按照软件的官网来就好了,记住遇到软件安装,上官网看文档就对了,和你安装qq没啥区别,只是放到linux系统里一样。
conda有详细介绍过
利用Anaconda在Windows及Linux下搭建Python环境【手把手版】
遇到问题咋办吗?
微软 Bing 搜索 - 国内版
搜狗微信搜索_订阅号及文章内容独家收录,一搜即达
Bioinformatics Answers
等等,实在不行了某度上搜也可以~
学完上面几步基本可以把生信软件串起来了,可以尝试拿开源数据试试了~
一定要开始做,光看没用
学门编程语言吧
python/perl/R生信常用。
上面拿到了软件输出的东西,如果要进一步为所欲为的处理(筛、删、统计啊、可视化展示等等),靠linux是不行的,必须学一个语言,老一辈用perl处理文本,用c写算法,现在好多都转python了(选什么看你的喜好和你所处的团队吧,团队一致即可);可视化,个人感觉生信领域R最强。
怎么学python
如果是一点都不会编程的话看,
《A Byte of Python》快速入门
中文版,LenKiMo/byte-of-python
英文版,swaroopch/byte-of-python
有点基础了,再看《Python编程:从入门到实践》,看前面几章就可以了

想精进:
Fluent Python
Python Cookbook 3rd Edition Documentation
《Effective Python:编写高质量Python代码的59个有效方法》
也可一看看小编的python学习笔记,参考书籍都在文末
详解Python数值、字符串处理pythonic生物人:Python可视化笔记43篇合集(建议收藏)
详解Python列表推导式|迭代器|生成器|匿名函数
详解pandas DataFrame, Series常用方法
详解Python模块、包、库
详解Python CONDA
详解Python字典、集合
详解Python模块化编程-自定义函数
详解Python好用的内置函数
小编的python可视化学习笔记

怎么学perl
Perl学习11之perl模块详解(二)
Perl学习15之perl读excel表格
Perl学习16之读文件,存入哈希,输出到文件
Perl学习19之生信简单运用(三)
等等。。。。
怎么学R?
你是如何自学R语言的?
生物信息初学者该如何系统性的掌握 R语言?
R语言基础(一)
R语言基础(2)-向量|矩阵|数组|数据框|数据IO|实用函数
R语言基础(3)-变量|运算符|变量简单操作
小编的R可视化学习笔记

灵活的构建分析流程
Snakemake - Snakemake 6.4.0 documentation
Nextflow’s documentation!
不得不学的统计学作为统计的博士生,你都读过哪些对你影响深远的统计书籍?
《中国卫生统计》
《生物医学研究的统计方法(第二版)》
想更精进生信
盘大牛生信软件的底层原理吧
多参加行业会议
多交流
多阅读前沿文献
推荐阅读
pythonic生物人:Python可视化笔记43篇合集(建议收藏)
pythonic生物人:从哪些方面着手提高Python?Python社区“老司机们”的这本书讲清楚了!
Python 从入门到精通推荐看哪些书籍呢?
你所读的统计学方向,有哪些不错的讲义(Notes)?
你是如何自学R语言的?
pythonic生物人:13个高清图助快速上手Python: NumPy/Pandas/SciPy/Matplotlib?
有哪些你看了以后大呼过瘾的数据分析书?
pythonic生物人:丰富的Python可视化“武器库”:从PyViz到Matplotlib
GitHub上都有哪些值得关注学习的R开源项目?
机器学习、数据科学 如何进阶成为大神?
作为一个研究生,有哪些你直呼好用的科研神器?
作为统计的博士生,你都读过哪些对你影响深远的统计书籍?
欢迎关注❤️:pythonic生物人
 新公网安备 65010402001845号
新公网安备 65010402001845号